综合《自然》杂志、《纽约时报》、《财富》杂志报道:11月30日,美国科技巨头谷歌旗下人工智能(AI)公司DeepMind宣布,他们研发的AlphaFold系统在最新一届国际蛋白质结构预测竞赛CASP14上击败所有对手,证明自己能够基于氨基酸序列,快速、精确预测蛋白质3D结构。这意味着该款AI系统,很大程度上解决了困扰全球科学家近50年的蛋白质折叠问题,将推动医学进步。AlphaFold已被用于解析新冠病毒。
绝大多数生理机能都与蛋白质息息相关,而每个蛋白质因自身氨基酸链折叠形成的独特3D结构,决定其具体作用。如何破解这一结构,就是蛋白质折叠问题。1972年,诺贝尔化学奖得主安芬森在获奖感言中提出著名假设,即“蛋白质的氨基酸序列应完全决定其结构”,引领了科学界此后近50年的研究方向。
安芬森假设面临的主要挑战,是蛋白质形成最终结构前,可能有无数种折叠方式。使用现有的X射线晶体学技术、低温电子显微镜等解析蛋白质结构,需花费数月、数年甚至数十年时间。而最新版AlphaFold系统,只需提供氨基酸序列,就可在数日内得出高度精确的预测结构。CASP联合创始人莫尔特教授表示,在很大程度上,蛋白质折叠问题得到了解决。
成功预测新冠蛋白结构
AlphaFold在蛋白质折叠问题上取得的突破,为生物学家打开了一扇新的大门。从胰岛素如何控制血糖水平,到抗体如何对抗新冠病毒,都由相应蛋白质结构决定。包括癌症在内的几乎所有疾病,都与细胞内蛋白质结构变化有关。因此,AlphaFold对蛋白质结构快速、精确的预测,将促进人类对细胞组成部分的理解,并对疾病预防和治疗、新药研发等带来重大影响。
冠状病毒的构成,包括可结合人体细胞受体的刺突糖蛋白、包裹病毒遗传物质的包膜蛋白、膜蛋白、核衣壳蛋白等。解析这些蛋白的3D结构,对了解新冠病毒致病机理及确定治疗方案具有重要意义。3月起,DeepMind就陆续发布AlphaFold对一些新冠病毒蛋白的结构预测,包括此前未知的ORF3a蛋白。在CASP14比赛中,该系统又预测了另一个新冠病毒蛋白ORF8的结构。该系统对这两个蛋白的预测均被证实相当准确。
平均误差仅为0.16纳米
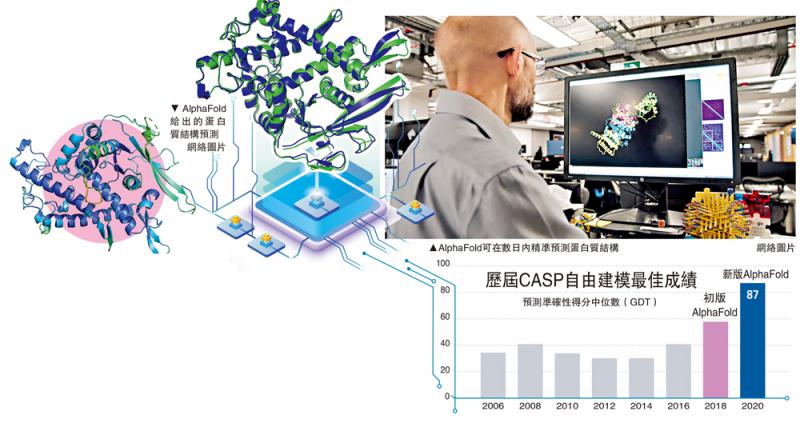
在自然界,由氨基酸链组成的蛋白质,可自发折叠成无数令人难以想像的形状。为解析这一过程,DeepMind研究人员使用包含约17万个蛋白质序列及其结构的公共数据库,对AlphaFold算法进行训练。2018年,初版AlphaFold参加CASP13并获得优胜。今年,新版AlphaFold在CASP14击败来自全球的约100个团队,使蛋白质结构预测达到前所未有的精确度。
CASP衡量预测准确性的主要指标是全局距离测试(GDT)得分,范围从0至100。新版AlphaFold总体得分中位数高达92.4,意味着预测平均误差仅为0.16纳米,相当于一个原子的直径。在测试给出的近100个蛋白靶点中,该系统对三分之二靶点给出的预测结构,与通过传统实验手段得到的相差无几。即便是在难度最高的自由建模分类中,该系统得分中位数也高达87,刷新2018年初版创下的近60分的纪录。在AlphaFold问世之前,各种方式的准确性得分中位数从未超过50。但DeepMind公司承认系统仍有很多预测不准的情况,需要继续改进。